最新 | 单细胞数据降维和可视化的优势工具--UMAP
高通量单细胞组学数据的一个显著性特点就是数据量大,一次能反映的细胞数量多。因此,通过降维和可视化去展示细胞数据特征是一个非常重要的工作。翻开各类发表的单细胞组学文章,不管是CNS的还是其他,几乎所有的结果中,映入眼帘的第一张图片通常是数据结果的降维图形化展示。

图1 PBMC细胞单细胞转录组数据展示图
实现高维数据可视化的理论基础是基于降维算法。降维算法一般分为两类:那些寻求在数据中保存距离结构的,以及倾向于保存局部距离而不是全局距离的。PCA[1]、MDS[2]等算法属于前者,t-SNE[3,4]、diffusion maps[5]等算法都属于后者。
对高维单细胞数据的可视化展示,以t-SNE为代表的非线性降维技术,由于其能够避免集群表示的过度拥挤,在重叠区域上能表示出不同的集群而被广泛运用。然而,任何技术方法都不是完美的,t-SNE也一样,它的局限性体现在丢失大规模信息(集群间关系)、计算时间较慢以及无法有效地表示非常大的数据集[6]等方面。
那么,有没有其它方法能在一定程度上克服这些弱点呢?UMAP就是这样一个能解决这些问题的降维和可视化的工具。
统一流形逼近与投影(UMAP,Uniform Manifold Approximation and Projection)是一种新的降维流形学习技术。UMAP是建立在黎曼几何和代数拓扑理论框架上的。UMAP是一种非常有效的可视化和可伸缩降维算法。在可视化质量方面,UMAP算法与t-SNE具有竞争优势,但是它保留了更多全局结构、具有优越的运行性能、更好的可扩展性。此外,UMAP对嵌入维数没有计算限制,这使得它可以作为机器学习的通用维数约简技术。
从上述定义可以看到,UMAP对于单细胞这类大数据、高维数据来说,是一个正中下怀的好工具。那么,在真实数据运用中,这种优势能体现出来吗?以下我们通过实际数据对比进行展示。
1.小数据集中,t-SNE和UMAP差别不是很大
我们分别以200个和500个单细胞的转录组数据为基础,分别通过t-SNE和UMAP进行降维可视化展示,得到如下图:
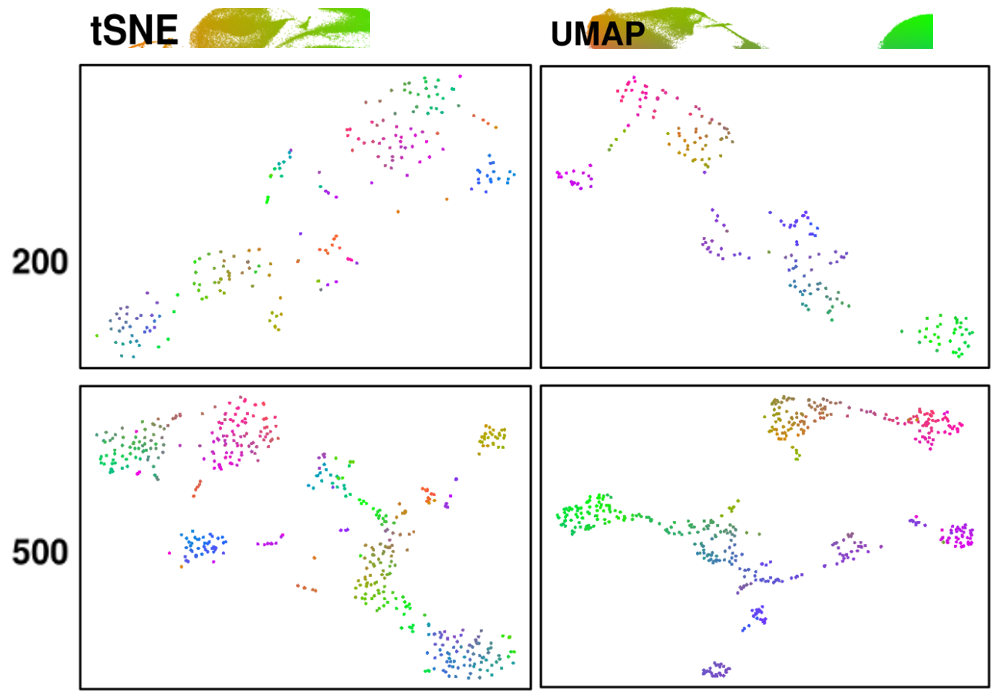
图2 200和500个细胞的单细胞转录组数据的t-SNE和UMAP降维分群图
可以看到,在小数据集下,两者对细胞集群的展示上,效果比较类似,基本能把不同类型的细胞群进行区分。所以,当数据集比较小的情景下,t-SNE已能足够展示不同群体细胞特征。
2.大数据集中,UMAP优势明显(30多万个细胞的降维可视化分析)
我们同时在一个数据集上运行PCA、UMAP和t - SNE,该数据集涵盖了来自8个不同的富含T和自然杀伤(NK)细胞的人体组织的35个样本,共含有30万多个细胞。

图3 PCA,t-SNE和UMPA的降维展示图
通过数据降维和可视化展示可以看出,PCA分群效果最差,UMAP和t-SNE都成功将与相似细胞群相对应的簇聚集在一起。但是与t-SNE相比,UMAP还提供了有用的和直观的特性、保留了更多的全局结构,特别是细胞子集的连续性。
接下来,我们对上述数据分别用细胞类型和组织来源类型进行着色展示:
(1)细胞类型着色

图4 根据细胞类型的细胞着色图
(2)组织类型着色

图5 根据组织来源的细胞着色图
通过在UMAP和t-SNE图上对来源组织进行颜色编码,我们发现t-SNE比UMAP更经常地根据来源组织分离细胞群;相反,UMAP根据细胞在每个主要集群中的起源对细胞进行排序,大致是从脐带血(CB)和外周血单个核细胞(PBMC)到肝脏和脾脏,以及一端的扁桃体到另一端的皮肤、肠道和肺。
当我们对特定T细胞标志性marker进行绘图,我们观察到UMAP能够概括T细胞内每个主要集群的分化阶段,从UMAP投影上的驻留记忆T细胞标志物CD69和CD103、记忆T细胞标记CD45RO和幼稚T细胞标记CCR7的表达水平可见。相比之下,虽然t-SNE在群集中确定了类似的连续性,但它们沿共同轴没有明显的结构。
由此可见,UMAP在大数据降维和可视化方面优势突出。

图6 Marker基因的细胞标记图
随着研究成本的降低和深入,单细胞研究的细胞数量和数据信息肯定会往上走,未来单细胞组学数据将会从现有千万的基础上增加2到3个数量级。因此,在这种发展趋势下,UMAP的发展空间会更大更广。
参考文献
[1]. Harold Hotelling. Analysis of a complex of statistical variables into principal components. Journal of educational psychology, 24(6):417, 1933.
[2]. J. B. Kruskal. Multidimensional scaling by optimizing goodness of fit to anonmetric hypothesis. Psychometrika, 29(1):1–27, Mar 1964.
[3]. Laurens van der Maaten and Georey Hinton. Visualizing data using t-sne.Journal of machine learning research, 9(Nov):2579–2605, 2008.
[4]. Laurens van der Maaten. Accelerating t-sne using tree-based algorithms.Journal of machine learning research, 15(1):3221–3245, 2014.
[5]. Ronald R Coifman and Stephane Lafon. Diffusion maps. Applied and computational harmonic analysis, 21(1):5–30, 2006.
[6]. Van Der Maaten, L. & Hinton, G. Visualizing high-dimensional data using t-SNE.journal of machine learning research. J. Mach. Learn. Res. 9, 26 (2008).
[7]. McInnes, L. & Healy, J. UMAP: uniform manifold approximation and projection for dimension reduction. Preprint at https://arxiv.org/abs/1802.03426 (2018).
新闻中心
News Senter
上海生物芯片有限公司
Shanghai Biochip Co., Ltd.
版权所有©上海生物芯片有限公司
电子邮箱:
marketing@shbiochip.com
地址: 上海市浦东新区张江高科技园区李冰路151号
技术电话:
4001002131
扫描查看
微信公众号

