单细胞分析雕刻师--常见整合方法比较(一)
—— Preface ——
随着单细胞测序技术的迅猛发展和市场的不断下沉,越来越多的研究人员都青睐于使用该技术来阐明一些生物学或医学问题,使通过传统bulk-RNA测序无法解决的事情得以实现成为可能,如细胞图谱的绘制、稀有细胞的鉴定与识别、细胞发育/分化轨迹的构建、肿瘤的精细化研究等。与此同时,也产生了海量的单细胞数据,而这些数据通常来源于不同的实验室,具有不同的构建时间、不同的操作人员以及不同的试剂批次等等。上述差异往往会对数据的合并造成严重的影响,导致批次效应的出现,进而干扰对真实的生物学效应的鉴别,因此,如何将不同来源的数据完美地系在一起一直是一个复杂的、具有挑战性的问题。在过去的十几年间,有数十种数据整合方法相继被开发出来,它们基于不同的原理或应用场景实现对数据的合并,在保留生物学差异的同时尽可能地去除批次效应。这里,我们选择了一些比较常见的工具或方法,包含ComBat、BBKNN、Seurat CCA、Seurat RPCA、Harmony、LIGER、fastMNN、Conos、Scanorama总共9种,通过应用于同一套数据对其进行比较。
—— Results ——
01 Preprocessing
对于每一个独立的样本,我们遵循 Seurat 标准分析流程进行预处理,并且依据已知的Marker基因和SingleR[1] 预测的细胞类型结果对其进行注释,然后将所有数据合并在一起,分别保存为RDS和Anndata对象以供后续使用。以其中一例样本进行演示 (10x, pbmc_1k)。读取数据创建Seurat对象,过滤表达基因数低于200、线粒体基因占比高于5%的细胞;然后对表达矩阵做标准化处理,结合高变基因(HVGs, High Variable Genes)使用PCA降维,选取前20个主成分(PCs, Principal Components) 进行后续的聚类及可视化,并使用SingleR对细胞进行注释;最后,结合已知的细胞特异性Marker进行注释的校验 (Figure1)。
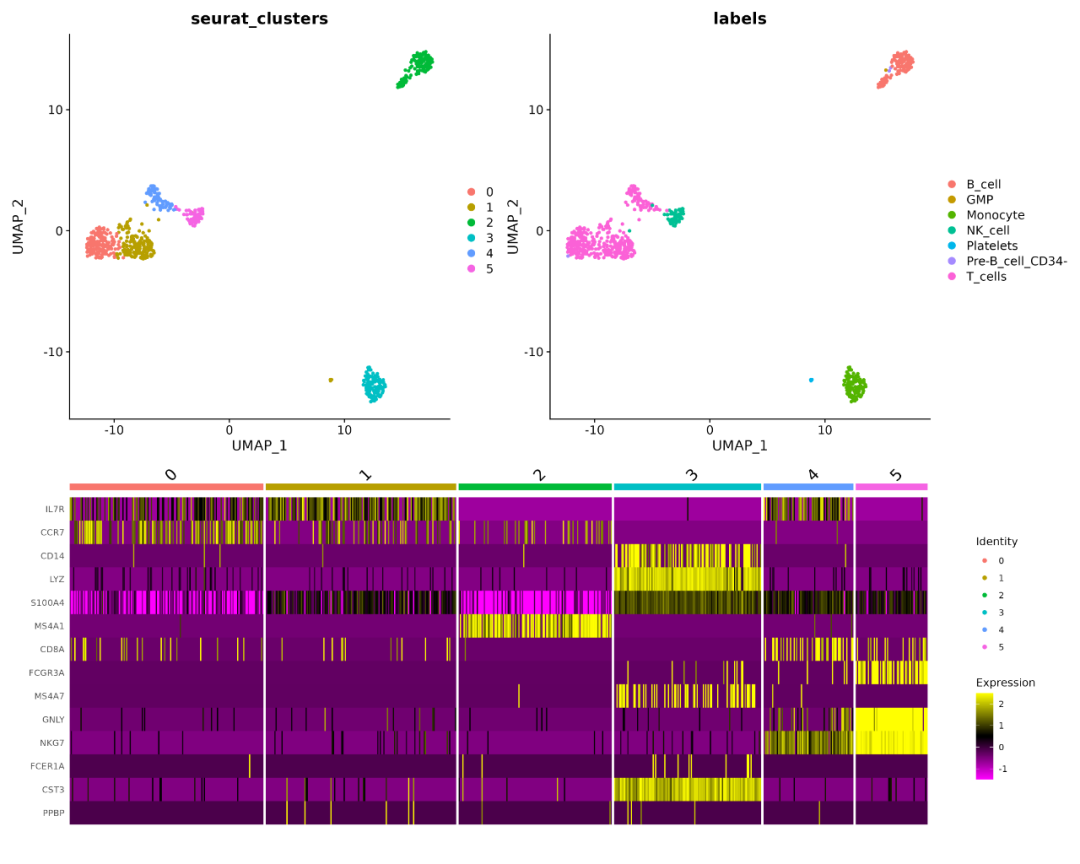
Figure1 | pbmc_1k样本聚类结果。上方图片为按照聚类结果和注释结果分组的UMAP降维图形,下图为Marker基因的表达热图,列按照聚类结果分组
02 unIntegration
按照如上方法对每个样本进行单独处理,然后合并所有样本。首先,我们在未矫正的情况下直接对数据进行分析,并按照不同的标签进行分组,如数据来源、细胞类型等 (Figure2)。其中,为了方便理解及简化后续分析,我们将细分的细胞类型简单合并,例如 pre-B_cell_CD34- , pro-B_cell_CD34+ 和 B cell 统称为B细胞。
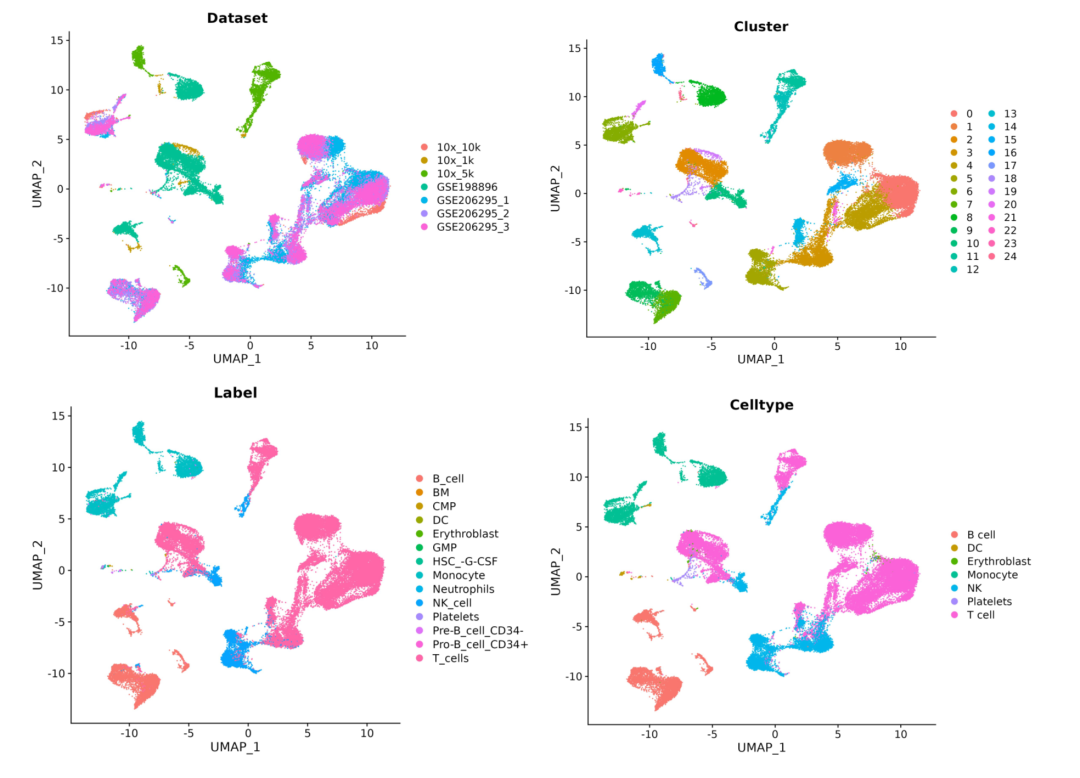
Figure2 | 未整合数据分布。四张子图分别为按照数据来源、聚类结果、细分细胞类型、合并细胞类型分组的UMAP图形展示
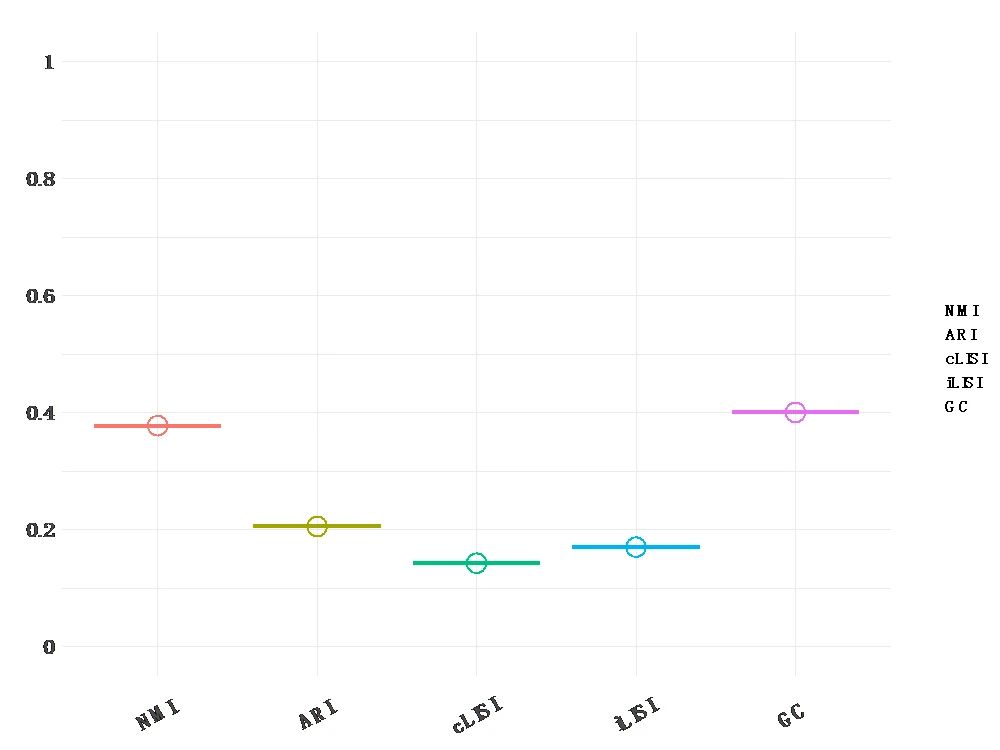
Figure3 | 未整合数据评分
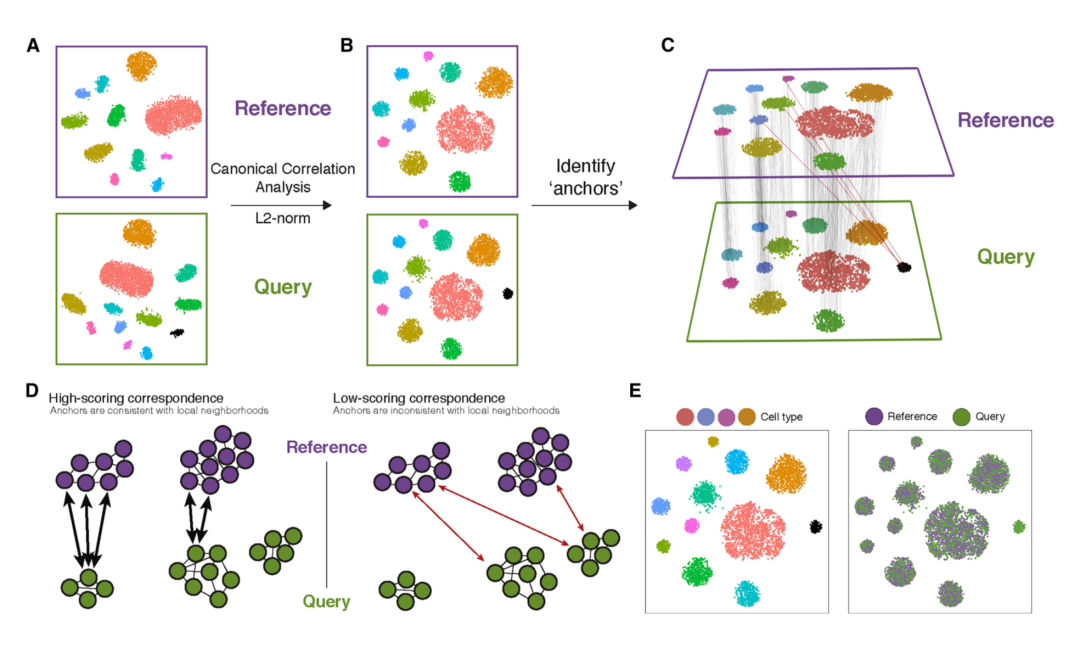
Figure4 | Seurat整合流程模式图。参考数据和查询数据共享一组类似细胞,通过CCA及L2正则化得到相关向量,映射在共享亚空间中,在该空间内鉴定MNN细胞对并且赋分,最后计算每个细胞的矫正向量用于转换表达数据
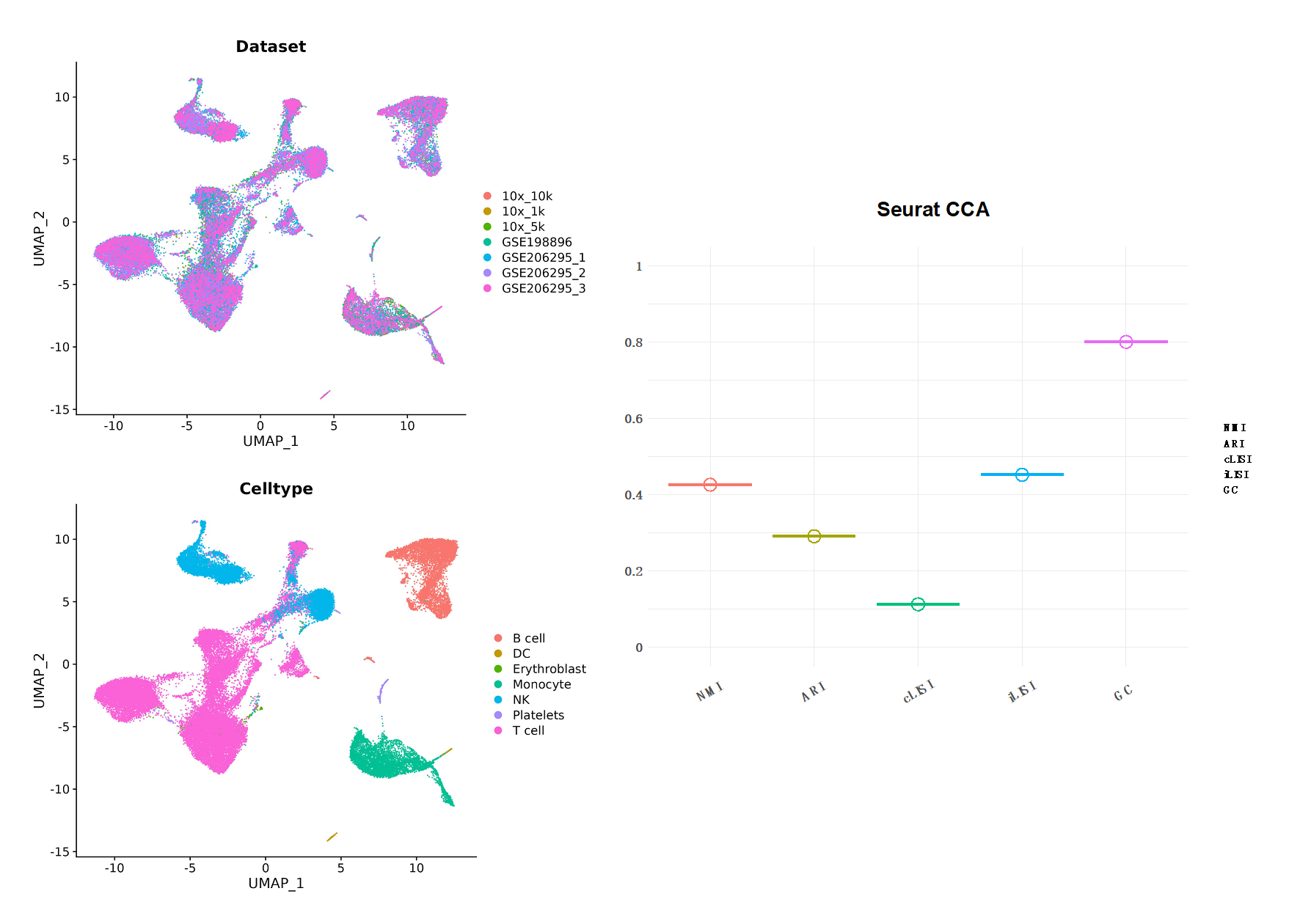
Figure5 | Seurat-CCA 整合分析结果。左边为UMAP降维图形展示,分别以数据集和细胞类型分组;右图是结果评分
04 Seurat RPCA
接下来我们使用Seurat中另一种用于数据整合的方法——RPCA(Reciprocal PCA),该方法使用RPCA而非CCA来识别数据集锚点。使用该方法确定任意数据集之间的锚点时,会将每个数据集投影到其他PCA空间中。在分析上,Seurat-RPCA和Seurat-CCA并无太大区别。
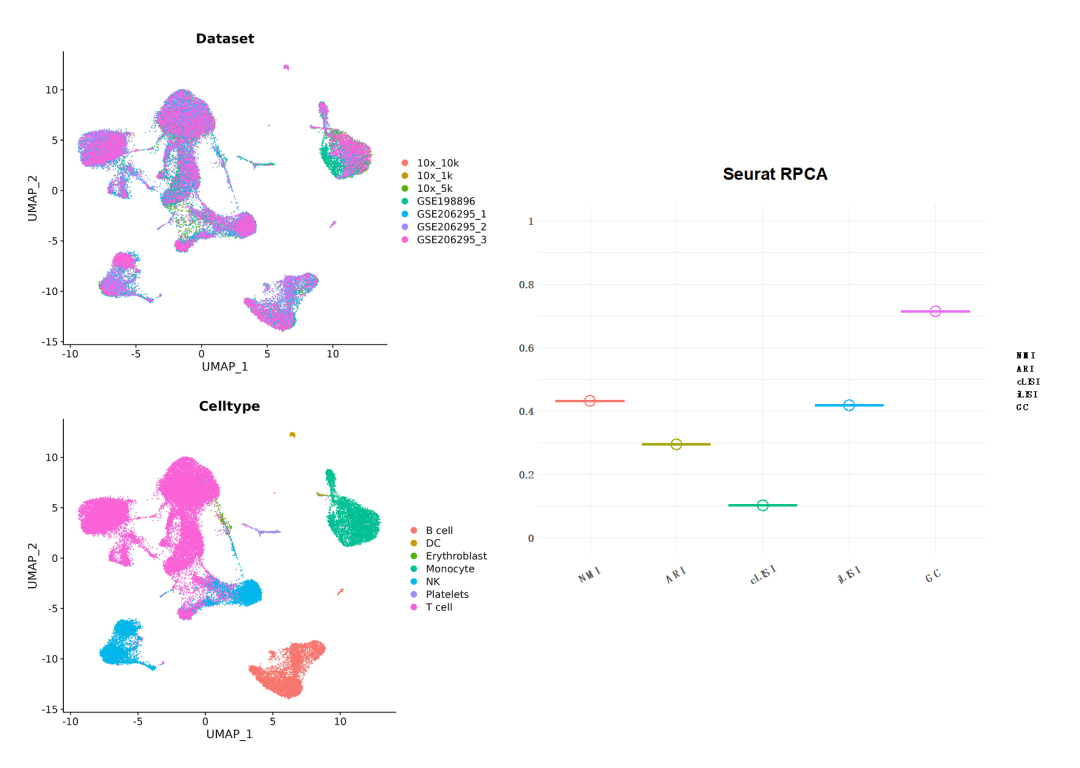
Figure6 | Seurat-RPCA 整合分析结果。左边为UMAP降维图形展示,分别以数据集和细胞类型分组;右图是结果评分
新闻中心
News Senter
上海生物芯片有限公司
Shanghai Biochip Co., Ltd.
版权所有©上海生物芯片有限公司
电子邮箱:
marketing@shbiochip.com
地址: 上海市浦东新区张江高科技园区李冰路151号
技术电话:
4001002131
扫描查看
微信公众号

